受精卵のエピゲノム編集に成功 – ゲノムに記された「付箋」を書き替える新手法
ひとつのゲノムから多彩な細胞がつくられるしくみ
私たちの身体はさまざまな種類の細胞や組織から構成されていますが、これらのほとんどの細胞が同じ遺伝情報(ゲノム)を核内にもっています。同じ遺伝情報をもつにも関わらず、どのようにさまざまな種類の組織や細胞がつくられるのでしょうか? これは細胞内において、「必要な遺伝子が読み出されること」「不必要な遺伝子の読み出しを制限すること」によって成し遂げられています。言い換えれば一冊の設計図(遺伝情報)を読み出す付箋の組み合わせによって、さまざまな細胞がつくられているのです。
この付箋の役割を果たすのがエピゲノムと呼ばれるDNAやヒストンなどに生じる化学修飾です。なかでもDNAメチル化は4つのDNA塩基、アデニン(A)、チミン(T)、グアニン(G)、シトシン(C)のうち、シトシンが受ける化学修飾であり、遺伝子発現の抑制、刷り込み遺伝子の発現制御(ゲノムインプリンティング)、X染色体の不活性化、レトロトランスポゾンなど動く遺伝子の転移抑制など、正常な個体発生において重要な役割を果たしていることが知られています。また、癌やさまざまな疾患においてもDNAメチル化情報の乱れが報告されており、細胞が正常な機能を発揮するためには正常なDNAメチル化パターンの形成が重要であることがわかっています。
付箋の意味を知る
細胞内でエピゲノムが形成・維持される過程においては、化学修飾を書き込む分子、消去する分子、認識する分子が協調して動くことが重要ですが、これらエピゲノムの形成に関わる分子はゲノムの広範囲に渡って機能しているため、ノックアウトの解析手法では特定ゲノム部位のDNAメチル化状態を操作することや、その部位におけるDNAメチル化が果たす生物学的な役割を解析することは難しいという問題も抱えていました。
実際、マウスにおいてはDnmtと呼ばれるDNAメチル化を書き込む分子、Tetと呼ばれるDNAメチル化を消去させる分子が存在していますが、いずれの分子の遺伝子破壊動物においても胎生致死や出生後の死亡あるいは生体に何かしらの異常をもって生まれることが知られています。このような課題をクリアするため、私たちはまずDNAメチル化をゲノムの部位特異的に書き込む手法の開発に取り組むことにしました。
受精卵でのDNAメチル化誘導
私たちは、任意の塩基配列に対するDNA結合モジュールを設計することが可能なゲノム編集と呼ばれる技術に着目しました。ゲノム編集で用いられているTALENおよびCRISPR/Cas9のDNA結合モジュールと、スピロプラズマと呼ばれる細菌が保有するDNAメチル化酵素のひとつであるSssIとの融合遺伝子を作製し、この人工遺伝子を細胞内に導入することでマウスのペリセントロメアに存在するDNA配列であるメジャーサテライトに対してDNAメチル化導入が可能か検討しました。
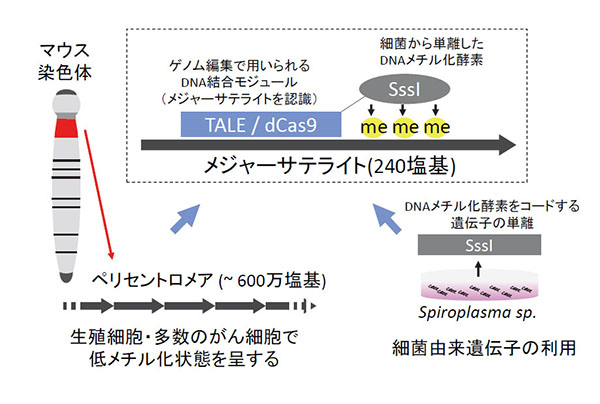
その結果、ペリセントロメアDNAが低メチル化を示すマウス受精卵や、DNAメチル化酵素を欠損したゲノムワイドにDNA低メチル化状態を示すES細胞のメジャーサテライトに、DNAメチル化を効率的に導入することが可能であることを示しました。このDNAメチル化の亢進はバイサルファイトシーケンスによる1塩基レベルの解像度だけでなく、DNAメチル化の生細胞レポータープローブ(mCherry-MBD-NLS)を用いた蛍光顕微鏡レベルでも検出可能なものであり、大規模にDNAメチル化が導入される様子をライブセルイメージングによって追跡することが可能でした。
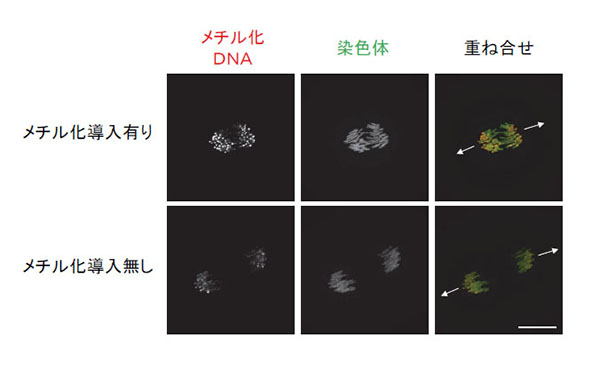
受精卵における分裂後期の染色体のスナップショット。「メチル化導入有り」の受精卵において分配方向の染色体末端近くにDNAメチル化の亢進が認められる。「メチル化導入無し」の染色体においても末端部分にメチル化DNAのシグナルが認められるが、これは元々細胞が持っているDNAメチル化状態を示している。矢印:染色体分配方向、スケールバー:20μm
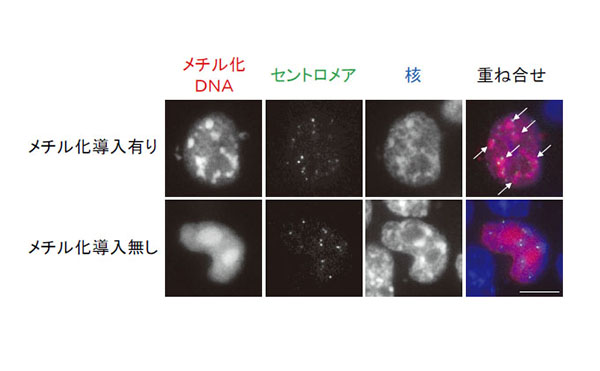
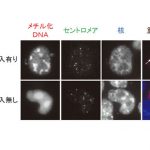
「メチル化導入有り」の細胞では核内のセントロメア近傍においてメチル化DNAのシグナルの亢進が認められる(矢印)。一方で、「メチル化導入無し」の細胞ではメチル化DNAのシグナルは核内でほぼ均一のシグナルとなる(核小体にシグナルが集積する)。スケールバー:20μm
さらに、ペリセントロメアにDNAメチル化を導入したマウス受精卵の細胞分裂期における染色体分配異常を調べたところ、DNAメチル化導入した受精卵とDNAメチル化導入していない受精卵において大きな違いは認められず、着床前初期胚発生のキネトコア機能にDNAメチル化は重要ではないことが示されました。従来のエピゲノム編集で使用されてきた哺乳動物由来のDNAメチル化酵素は、協調して働く分子を必要としますが、本研究で作製された細菌由来の人工酵素はそれ単独で機能するため、他の生体作用の影響を受けずに安定したDNAメチル化導入効果が期待されます。
今後の展開
受精卵を含むマウス生殖細胞のセントロメア(細胞分裂に必須な染色体配列)およびペリセントロメアでは、体細胞と比べて大規模なDNA脱メチル化が生じていることが報告されていますがその具体的な意義については明らかにされていません。本研究で開発した手法はこの疑問に答える手段のひとつになると考えています。また、複数のがん細胞においても生殖細胞と同様にセントロメアのDNA脱メチル化が大規模に生じていることが報告されており、がんに特徴的なゲノム不安定性との相関性が指摘されています。本研究によるDNAメチル化操作によって、がん細胞におけるゲノム不安定性とDNA脱メチル化状態との因果関係を研究することが可能となり、新たながん研究の解析ツールとなることが期待されます。
また、本研究で使用したDNA結合モジュールは任意のゲノム領域に設計できることから、ペリセントロメア以外のさまざまなゲノム部位に対してDNAメチル化の導入を行うことが可能だと考えられます。遺伝子破壊を伴わずに遺伝子の発現抑制を行うことによって、がん遺伝子をはじめとする疾患原因遺伝子の発現抑制など、ゲノムを書き換えない遺伝子治療への将来的な応用展開が期待されます。
参考文献
Yamazaki T, Yamagata K, Baba T. (2007). Time-lapse and retrospective analysis of DNA methylation in mouse preimplantation embryos by live cell imaging. Dev Biol. 304, 409-419.
Yamagata K, Yamazaki T, Miki H, Ogonuki N, Inoue K, Ogura A, Baba T. (2007). Centromeric DNA hypomethylation as an epigenetic signature discriminates between germ and somatic cell lineages. Dev Biol. 312, 419-426.
Yamazaki T, Hatano Y Handa T, Kato S, Hoida K, Yamamura R, Fukuyama T, Uematsu T, Kobayashi N, Kimura H, Yamagata K. (2017). Targeted DNA methylation in pericentromeres with genome editing-based artificial DNA methyltransferase. PLoS One. 12: e0177764.
この記事を書いた人

-
北里大学メディカルセンター研究部門上級研究員
明治大学農学部卒業、筑波大学大学院生命環境科学研究科にて博士(農学)を取得後、学校法人北里研究所入所、2009年4月より現職。専門はエピジェネティクス。現在までDNAメチル化を「見る(プローブの開発)」、「操作する(エピゲノム編集)」研究に従事。今後はエピゲノム編集技術を生かした応用研究を展開していきたい。