遺伝子が転写されるときのヌクレオソームの”動き”を捉えた!
ヌクレオソームのふるまいとは?
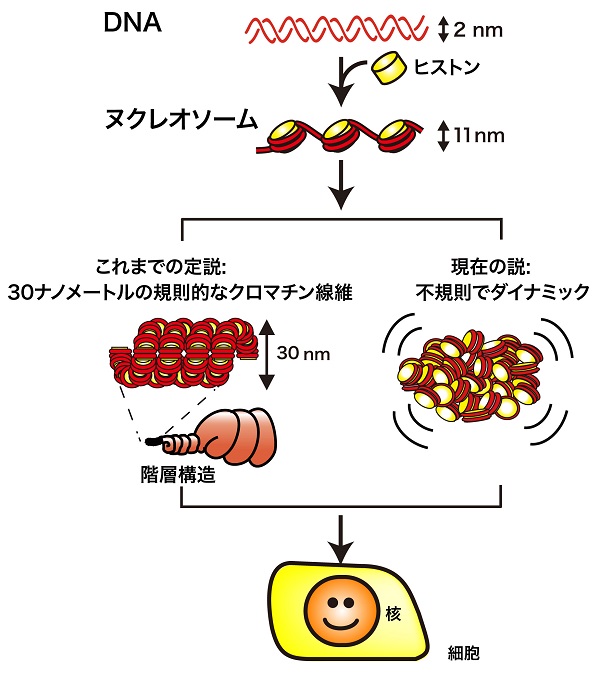
私たちの体は約40兆個の細胞から構成されています。それぞれの細胞には、生命の設計図とも称される全長約2mのヒトゲノムDNAが収納されています。このDNAは直径2nm(10-9m)のとても細い糸で、「ヒストン」とよばれる樽状のタンパク質に巻かれることで、直径約11nmの「ヌクレオソーム」を作っています。ヌクレオソームと、これに結合する他のタンパク質を合わせて「クロマチン」と呼びます。
長いあいだ、ヌクレオソームはらせん状に規則正しく折りたたまれ(クロマチン線維)、さらに巻かれて階層構造を作ると考えられてきました。しかし、私たちは2007年よりクライオ電子顕微鏡やX線散乱を用いた実験によって、定説のような規則正しい階層構造は存在せず、ヌクレオソームが不規則に、そしてダイナミックに組織化され、細胞内に収められていることを提唱してきました。
これ以降、生きた細胞内でのヌクレオソームのふるまいについて理解が求められてきましたが、ヌクレオソームは数が膨大でかつその動きは非常に小さく、従来の光学顕微鏡を用いて観察することは困難でした。
遺伝子が転写されるときのヌクレオソームの動きとは?
私たちの研究室では、超解像蛍光顕微鏡を駆使することで、個々のヌクレオソームのふるまいを、生きたままの細胞内で観察する「単一ヌクレオソームイメージング」に成功しました。
成功の鍵はふたつありました。ひとつは、細胞1個あたり3000万個程度存在するヌクレオソームを、低濃度の蛍光色素(テトラメチルローダミン; TMR)でまばらに標識し、光るヌクレオソームの数を制限したことです。もうひとつは、シート状の光で細胞核を照らすことで、光が横切る極薄い領域にあるヌクレオソームだけを光らせ、顕微鏡観察における背景光を劇的に抑えたことです。これらの工夫により、生きた細胞の中で個々のヌクレオソームを撮影し、その動きを詳細に調べることが可能となりました。
私たちは、撮影した個々のヌクレオソームの輝点を2次元ガウス関数でフィットし、ヌクレオソームの位置を高精度(15nm)で決定しました。さらに、個々のヌクレオソームの動きを追跡することで、一定時間におけるヌクレオソームの動きの大きさの指標である平均二乗変位も見積もりました。
次に、単一ヌクレオソームイメージングを用いて、「遺伝子が転写される際のヌクレオソームの動き」を解析しました。細胞をRNAポリメラーゼII(RNAPII)の阻害剤で処理して転写を阻害し、ヌクレオソームの動きを解析したところ、驚くべきことに、その投与量に依存してヌクレオソームの動きが活発になることがわかったのです。
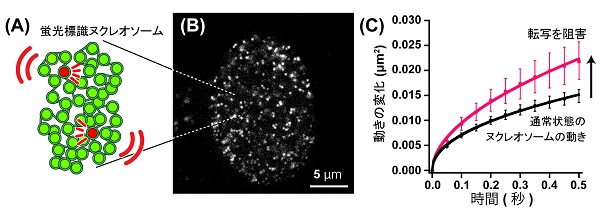
ここで、観察したヌクレオソームの動きを動画で見てみましょう。左側は通常状態の細胞内のヌクレオソームの動きで、ひとつひとつのドットがひとつひとつのヌクレオソームを示しています。右側は転写を阻害した際のヌクレオソームの動きで、左側の通常状態と比べてヌクレオソームの動きが活発化していることがわかります。
超解像蛍光顕微鏡による核内の単一ヌクレオソームの動きの動画(1コマ50ミリ秒)(Nagashima et al., 2019より)
複数の転写阻害剤を試しても、同様の結果が得られました。つまり、転写が起こる際、ヌクレオソームの動きが束縛されていることが明らかになったのです。一般に、転写されているクロマチン領域は、オープンでダイナミックであると考えられてきたので、この結果は予期せぬものでした。
一方、阻害剤処理には常にさまざまな間接効果のリスクがつきまといます。たとえば、ある種の転写阻害剤処理を長時間行うと核の内部構造が変化することが知られています。そこで、より直接的に「転写が阻害された状態のヌクレオソームの動き」を評価するため、国立遺伝学研究所・鐘巻将人教授らが考案したオーキシンデグロン(AID)法を用いて、RNAPIIの最も大きなサブユニットであるRPB1を急速に、そして特異的に分解除去しました。その結果、転写阻害剤処理を行ったときと同様にヌクレオソームの動きが顕著に上昇しました。「転写を阻害するとヌクレオソームの動きが上昇する」ことへのさらなる確証が得られたわけです。
クロマチンは「転写ハブ」を介してネットワークをつくる
RNAPIIによる転写を阻害すると、ヌクレオソームの動きが上昇することから、RNAPIIがヌクレオソームの動きを抑えていると思われます。このことから私たちは、クロマチンは転写が起こる場所(RNAPIIの集まったところ)を「ハブ」として、ゆるやかなネットワークを形成していると考えました。つまり、通常状態ではこのゆるやかなネットワークがクロマチンの動きをグルーバルに抑制しており、RNAPIIを阻害または除去すると、このネットワークが失われてクロマチンの動きが上昇するわけです。
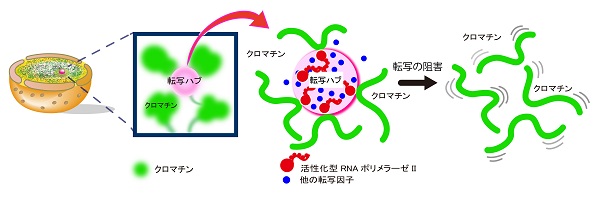
この考えを試すために、名古屋大学工学研究科・笹井理生教授グループの協力を得て、計算機シミュレーションによってクロマチン環境を再構築しました。RNAPIIの転写ハブのありなしで、クロマチンの動きをシミュレーションした結果、予測通りRNAPIIのハブを除くとクロマチンの動きは上昇しました。
そのシミュレーション結果を動画で見てみましょう。緑のボールはヌクレオソームの塊、ピンクのボールは転写ハブ、赤いボールは転写しているRNAポリメラーゼIIを表しています。転写ハブが存在しない場合は、転写が阻害された状況を示しており、転写ハブが存在する状況より緑のボールが激しく動いているのがわかります。
転写ハブが存在する状況でのクロマチンの動きの再現(名古屋大学工学研究科・笹井理生教授グループ提供、Nagashima et al., 2019より)
転写ハブが存在しない状況でのクロマチンの動きの再現(名古屋大学工学研究科・笹井理生教授グループ提供、Nagashima et al., 2019より)
この結果から、計算機シミュレーションによっても、クロマチンはRNAPIIが集まった場所をハブとしてゆるやかなネットワークを形成し、束縛されていることが支持されました。
転写とヌクレオソームの理解に向けて
今回私たちは、単一ヌクレオソームイメージングと計算機シミュレーションを用いて、生きたヒト細胞核のゲノムワイドなクロマチンの動きを調べました。その結果、RNAPIIによる転写を阻害するとヌクレオソームの動きが上昇することを見出しました。このことから、クロマチンは、RNAPIIが集まり転写が起こる場所をハブとしてゆるやかなネットワークを形成していると考えられます。このような転写のハブは、RNAPIIおよび転写関連タンパク質を局所的に高濃度に保ち、転写するクロマチン領域の動きを抑えることで転写効率の向上に寄与していると考えられます。
今回の私たちの知見は転写の分野で古くから提唱されてきた、ゲノムDNAが集まって転写が起こるという「転写ファクトリー」の概念とよく一致します。また最近、RNAPIIや転写因子が核内で局所的に集まって液滴を作り、転写を活発化するという仮説も提唱されています。転写のハブは液滴かもしれません。転写因子の液滴とヌクレオソームの動きの関係を明らかにすることは今後の課題といえるでしょう。
細胞の多くの活動は、ゲノムDNA上の遺伝情報を読み出す転写から始まります。近年、転写の異常によって、ガン化・細胞死、さらには分化異常など、さまざまな細胞障害をもたらすことがわかってきました。本研究の成果によって、このような細胞の異常や関連疾患の理解が進むことも期待されます。
参考文献
・Nagashima, R., Hibino, K., Ashwin, S.S., Babokhov, M., Fujishiro, S., Imai, R., Nozaki, T., Tamura, S., Tani, T., Kimura, H., Shribak, M., Kanemaki, M.T., Sasai, M., Maeshima, K. “Single nucleosome imaging reveals loose genome chromatin networks via active RNA polymerase II” Journal of Cell Biology, 218, 1511-1530. (2019)
・Maeshima, K., Ide, S., Babokhov, M. “Dynamic chromatin organization without the 30-nm fiber” Current Opinion in Cell Biology, 58, 95-104 (2019)
・Nozaki, T., Imai, R., Tanbo, M., Nagashima, R., Tamura, S., Tani, T., Joti, Y., Tomita, M., Hibino, K., Wendt, K.S., Okada, Y., Nagai, T., Maeshima, K. “Dynamic organization of chromatin domains revealed by super-resolution live-cell imaging” Molecular Cell, 67, 282-293. (2017)
この記事を書いた人

-
佐藤 か奈(さとう かな)
東京理科大学基礎工学部生物工学科在学中。
日比野 佳代(ひびの かよ)/写真左
国立遺伝学研究所助教。大阪大学大学院修了、学振特別研究員、理化学研究所、理化学研究所生命システム研究センター(QBiC)の研究員を経て、2015年から現職。
kayo_hibino@nig.ac.jp
前島 一博(まえしま かずひろ)/写真右
国立遺伝学研究所教授。大阪大学大学院修了、スイスジュネーブ大研究員、理研専任研究員を経て2009年から現職。遺伝情報が検索され読み出される原理を追求中。いっしょに興味を持って追求してくれる方、募集中!
kmaeshim@nig.ac.jp
http://maeshima-lab.sakura.ne.jp
この投稿者の最近の記事
 研究成果2019年5月16日遺伝子が転写されるときのヌクレオソームの”動き”を捉えた!
研究成果2019年5月16日遺伝子が転写されるときのヌクレオソームの”動き”を捉えた!



