細胞1個でDNA複製を全ゲノム解析できる時代が来た! – DNA複製からゲノム制御の仕組みを探る
ゲノムDNAの複製タイミングとは?
生物が持つ基本的な性質のひとつは、増えることです。その構成要素である細胞は分裂を繰り返すことで数が増えていきますが、分裂前には必ずゲノムDNAが倍加(複製)されなければなりません。ゲノムDNAが過不足なく正確に複製されることは、細胞の持つ遺伝情報が世代を経ても正しく維持されるために必要不可欠です。
このゲノムDNA複製の際には、複製される順序(複製タイミング)が制御されていることが知られています。一般的に多細胞生物では、転写されやすいユークロマチン領域が細胞周期のS期前半に、転写されにくいヘテロクロマチン領域がS期後半に複製されることが知られています。
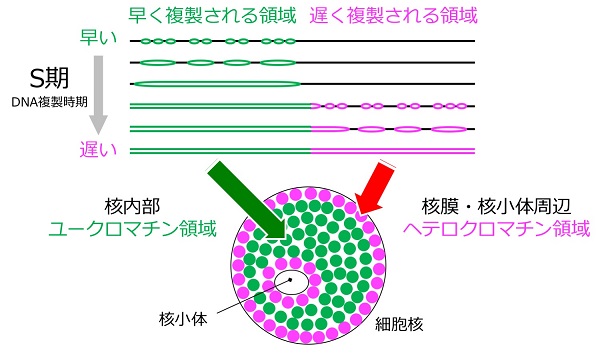
ゲノム上でS期の早い時期に複製される領域(緑色)は核の内部に存在し、遺伝子発現の活発なユークロマチン領域に相当する。遅く複製される領域(ピンク色)は核膜や核小体周辺に局在し、遺伝子発現が抑制されているヘテロクロマチン領域に相当する。
ここで複製タイミング制御の重要性がわかりやすい例をひとつ紹介します。
哺乳類の雌の体細胞では、2本のX染色体のうち片親由来の1本のX染色体が不活性化されています。不活性化されたX染色体に存在する大部分の遺伝子は発現が抑制されており、この染色体は全域がS期後半に複製されますが、これは哺乳類に共通に見られる特徴です。雌の初期発生には正常なX染色体の不活性化が必須であることから、このプロセスに関与する複製タイミングの制御は哺乳類の発生に重要であると考えられます。
1細胞レベルでゲノムDNAの複製タイミングを調べるには?
では、ゲノムDNAの複製タイミングはどのように解析するのでしょうか? ゲノムDNAのどの領域がS期のいつ複製するのかを、塩基配列レベルで全ゲノム解析できるようになったのは、2000年代に入ってからです。
平谷らは2008年に、高解像度のDNAマイクロアレイを用いた、マウスの全ゲノムDNA複製タイミング解析法を確立しました。複製された領域をラベルするために、ゲノムDNAにBrdU(塩基類似体)を短時間取り込ませた細胞を固定し、S期前半と後半の細胞をそれぞれ回収し、抗BrdU抗体でそれぞれの細胞集団で複製されたゲノム領域を濃縮して、マイクロアレイもしくは次世代シーケンサーを用いて全ゲノム解析する、というものです。
これは、現在も行われている最もスタンダードな全ゲノムDNAの複製タイミング解析法ですが、数万個の細胞を使う必要があるため、この解析から得られた複製に関する知見は、実は数万個の細胞の平均像に過ぎませんでした。個々の細胞で実際にどのように複製が進行するのか、つまり個々の細胞において複製タイミングがどう制御されているのかは、分子生物学における未解決の問題でした。
そこで今回我々は、1個の細胞からDNA複製を全ゲノム解析する手法の開発に取り組みました。S期の細胞1個ずつからゲノムDNAを取り出し、各ゲノム領域間のコピー数の差を検出できれば、複製前(1コピー)と複製後(2コピー)の領域を識別できる可能性があると我々は考えました。
この考えをもとに我々は新しい方法論の開発に取り組み、ゲノムDNAが複製される様子を1細胞レベルの全ゲノム解析によって捉える「scRepli-seq(single-cell DNA Replication sequencing)法」の開発に成功しました。
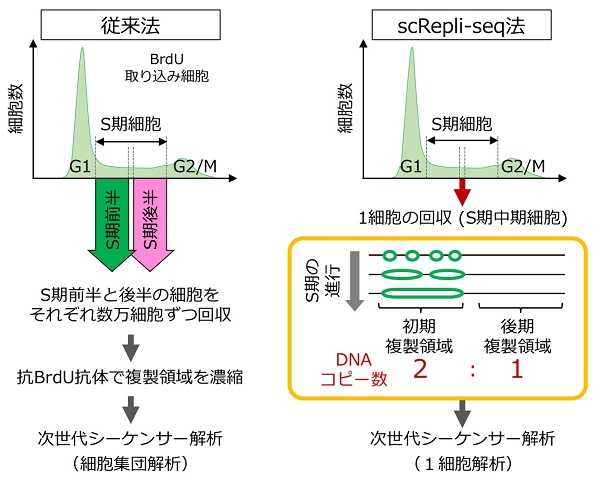
従来法では抗BrdU抗体による免疫沈降と呼ばれる濃縮作業を行うため、数万個の細胞が必要である。一方、scRepli-seq法ではS期細胞を1個ずつ回収する。各々の細胞から抽出したゲノムDNAの全ゲノム増幅を行い、次世代シーケンサーを用いてこのゲノムDNAを解読する。S期中期の細胞を用いて、複製前の領域はDNAコピー数が1、複製後の領域はDNAコピー数が2であることを利用すると、初期複製領域(複製後)と後期複製領域(複製前)を識別することが可能となる。
DNAの複製プロセスは細胞間で高度に保存されていた
我々はこの手法をマウスとヒトの細胞に適用し、さらに一塩基多型を用いて、父方と母方由来の染色体(相同染色体)を識別したscRepli-seq解析を実現し、細胞中の染色体1本1本がまさに複製されていく様子を捉えました。
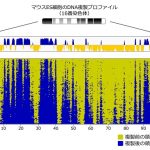
マウスES細胞を用いたscRepli-seq解析の結果を詳しく見てみると、細胞間のDNA複製プロファイルのゆらぎ(ばらつき)は予想よりも遥かに小さく、ゲノムDNA複製のプロセスは細胞間で高度に保存されていることがわかりました。ES細胞では複数の遺伝子の発現が細胞間でばらついていることが知られていますが、対照的にDNA複製タイミングは細胞間で安定であるという興味深い知見です。
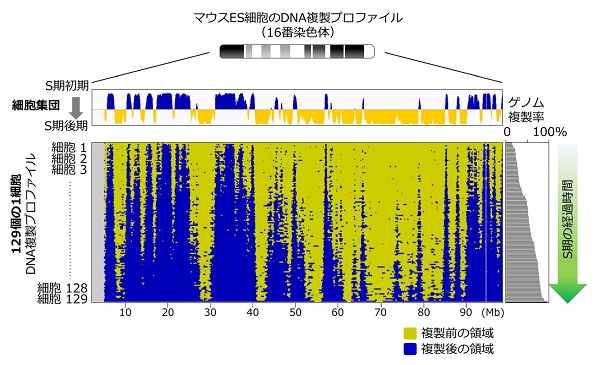
細胞は、S期全域から回収し、ゲノムDNA複製率に従って並べた(ほとんど未複製なものから複製完了に近いものまで)。細胞集団の平均像として見えていたゲノムDNA複製のプロセスが、個々の細胞のそれにかなり近いことがわかる。
また、マウスES細胞を分化させたところ、分化後にDNA複製プロファイルは大きく変わるものの、分化後も細胞集団中のゆらぎは依然小さいことがわかりました。以上の結果から、これまで数万個の細胞の平均像として見えていたゲノムDNA複製のプロセスは、個々の細胞のそれにかなり近いことが明らかになりました。
冒頭でも触れたように、DNA複製タイミングは染色体の高次構造ともよく相関していることが知られています。近年のHi-C法と呼ばれる全ゲノム解析を用いた染色体の三次元構造の研究から、間期の細胞核の内部で各々の染色体は、100万塩基対ほどの球状に折り畳まれたトポロジカルドメイン(TAD:Topologically Associating Domain)が数珠つながりになった構造をとると考えられています。
TADはさらに折り畳まれ、遺伝子がよく転写されているTADが集まって「Aコンパートメント」と呼ばれる領域を形成し、転写されていないTADが集まって「Bコンパートメント」と呼ばれる領域を核内空間に形成するとされています。
細胞集団を使った解析によると、ユークロマチンに相当するAコンパートメント領域はS期前半に複製される領域と、ヘテロクロマチンに相当するBコンパートメント領域はS期後半に複製される領域と非常によい一致を示しますが、今回のscRepli-seq解析で得られた1細胞全ゲノム複製プロファイルも、コンパートメントA/Bの分布と非常によく一致することがわかりました。
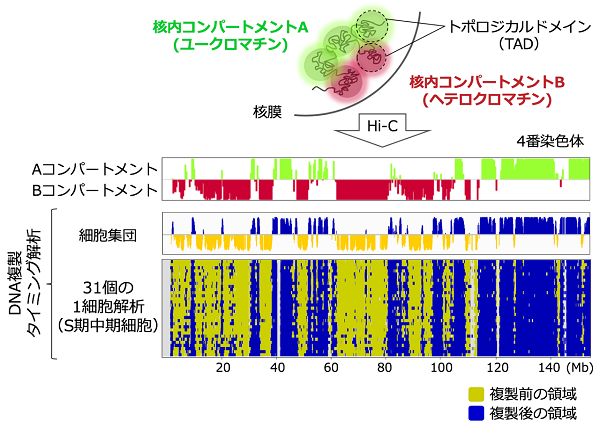
上段のAコンパートメント(緑)とBコンパートメント(赤)は、それぞれS期中期細胞における複製後(青色=S期前半に複製される領域)と複製前(黄色=S期後半に複製される領域)のゲノムDNA領域に対応していた。
個々の細胞間で複製プロファイルが高度に保存されているという前述の結果と併せると、核内コンパートメントA/Bの分布も細胞間で高度に保存されているという興味深い可能性が示唆されました。
細胞間で複製タイミングのゆらぎが大きい場所とは?
一方で、DNA複製時期が細胞間で比較的大きくゆらぐゲノム領域の存在も明らかになりました。我々は、ゆらぎやすい領域の特徴をいくつか明らかにしましたが、ここでは最も興味深いと感じた部分を紹介します。
ゲノムは、細胞種によらず複製時期が変わらない「不変領域」と、細胞分化に伴い複製時期が変わる「可変領域」に分類することができます。マウスではゲノムの約50%が可変領域に相当すると考えられています。
未分化のマウスES細胞をscRepli-seq解析したところ、細胞間で複製時期のゆらぎが有意に大きい領域として、分化後に複製時期が変わる可変領域が見出されました。一方で、細胞種によらず複製時期が変わらない不変領域のゆらぎは小さいものでした。また、分化後の細胞では、上記のどちらの領域においてもそのようなゆらぎは観察されませんでした。
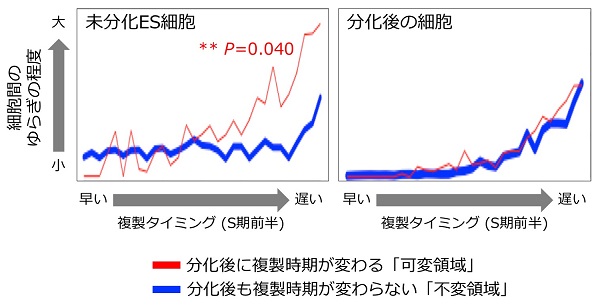
未分化ES細胞では分化後に複製時期が変わる可変領域で複製時期のゆらぎが有意に大きいのに対し、細胞種によらず複製時期が変わらない不変領域や分化後の細胞では、そのようなゆらぎは観察されなかった。
つまりこの結果は、未分化ES細胞の段階で将来どのゲノム領域の複製タイミングが変化するかが予測可能であることを示唆しています。胚発生初期の未分化細胞は、増殖を繰り返しながらさまざまな種類の器官や組織の細胞に姿や形を変えていきます(細胞分化)。前述の結果は、細胞分化に伴う複製タイミング・核内コンパートメント変化が、未分化細胞における可変領域の染色体高次構造の不安定性に起因しているかもしれないという興味深い可能性を示唆しています。
これは、今回我々が開発した細胞1個レベルの全ゲノムDNA複製解析であるscRepli-seq法によって初めて明らかになった現象であり、未だにほぼ手付かずの状態にある複製タイミングおよび核内コンパートメントの制御メカニズムを考えるうえで重要な発見であると考えています。
「scRepli-seq法」への今後の期待
我々はscRepli-seq法により、ゲノムDNAの複製タイミングが細胞間でほとんどゆらがないという普遍的な現象を見出しました。一方で、複製タイミングが比較的ゆらぐ領域の存在も明らかにしました。今回の結果から、複製タイミング制御やその生物学的意義の理解に一歩近づくことができたと思っています。
今回はヒトとマウスの細胞を使った解析を行いましたが、scRepli-seq法はDNAコピー数を検出するという極めて単純な原理に基づいているため、あらゆる生物種に適用することが可能です。また、これまで細胞数の問題から複製タイミングを調べることができなかった希少細胞での解析や、がん細胞のように不均一な細胞集団で個々の細胞の複製状態を知ることもできるようになります。そのため、将来的にはDNA複製異常や染色体構造異常を伴う疾患の理解にも貢献できると考えています。
今後も、scRepli-seq法を活用してDNA複製の仕組みを理解することによって、我々の遺伝情報の実体であるゲノム・染色体をより深く理解していきたいと考えています。また、scRepli-seq法は1細胞解析でありながら、実験は非常にシンプルで再現性良く解析できる手法なので、多くの研究者にscRepli-seq法を使っていただけたら嬉しく思います。
参考文献
Saori Takahashi, Hisashi Miura, Takahiro Shibata, Koji Nagao, Katsuzumi Okumura, Masato Ogata, Chikashi Obuse, Shin-ichiro Takebayashi, Ichiro Hiratani. “Genome-wide stability of the DNA replication program in single mammalian cells” Nature Genetics, 51, 529-540 (2019)
この記事を書いた人

-
高橋 沙央里(Saori Takahashi)/ 写真左
神戸・理化学研究所生命機能科学研究センター、基礎科学特別研究員(発生エピジェネティクス研究チーム)。2014年東京医科歯科大学大学院生命情報科学教育部修了(理学博士)。大阪大学蛋白質研究所でのポスドクを経て、2016年より現在の研究室でDNA複製タイミングに関する研究に従事。哺乳類の発生・分化におけるエピゲノム制御に興味があり、大学院時代から一貫してエピジェネティクス研究に携わっている。
平谷 伊智朗(Ichiro Hiratani)/ 写真右
神戸・理化学研究所生命機能科学研究センター、チームリーダー(発生エピジェネティクス研究チーム)。染色体三次元構造の発生制御の実態を独自の視点で解き明かしたいと考えています。